Senescencia celular
La senescencia celular es el proceso iniciado como respuesta al estrés y daño ocurrido en una célula, y constituye una ruta alternativa de respuesta a la muerte celular programada y es de vital importancia para suprimir la formación de células cancerosas. También está asociada a la reparación de tejidos e inflamación de los mismos, procesos asociados al crecimiento de tumores. De esta manera, la senescencia celular está asociada a los procesos de supresión y promoción de tumores simultáneamente, al igual que en el envejecimiento y reparación de tejidos, roles que son diametralmente opuestos; sin embargo, de seguir el comportamiento in vitro, este proceso podría considerarse un ejemplo de pleiotropía antagonística, donde un gen puede tener un impacto de adaptación positivo en algunos rasgos y simultáneamente un impacto negativo sobre otros.
Historia[editar]
Los procesos de senescencia celular fueron descritos por primera vez por Leonard Hayflick en su estudio sobre el crecimiento de fibroblastos humanos in vitro, donde encontró que en directa contravía de los postulados de Alexis Carrel (donde las células de mamíferos pueden crecer de manera infinita en cultivos in vitro), las células tienen un límite de crecimiento entre 40 y 60 ciclos de replicación[1]
El experimento consistía en mezclar dos cultivos de fibroblastos de diferentes "edades", un cultivo masculino que se encontraba en su cuadragésimo aumento al doble de población y uno femenino que está solo en el décimo. El cultivo mixto era comparado con dos cultivos de control sin mezclar, Hayflick y Morehead vieron que en el cultivo mixto, las células masculinas habían desaparecido después de un tiempo, y postularon que estas células podían "recordar" su edad a pesar de estar rodeadas de células jóvenes y se habían vuelto senescentes, con base exclusivamente en el número de veces que se habían replicado.[2]
En este primer artículo de Hayflick se pueden ver dos enunciados que serán fundamentales para el estudio futuro de la senescencia celular. En el primero declara que las únicas células somáticas que podrían escapar al proceso de senescencia son aquellas que han tomado las propiedades de células cancerígenas, y la segunda infiere la posibilidad de la senescencia celular en cultivos con el envejecimiento de seres vivos..
Definición e identificación de células senescentes[editar]
Existe una serie de características fisiológicas y moleculares que definen a una célula senescente y la diferencian de aquellas quiescentes y terminalmente diferenciadas. Las células entran en estado senescente principalmente en respuesta al daño oncogénico.
Estímulos de senescencia[editar]
Es importante recordar que la senescencia celular es un proceso iniciado por distintos estímulos, y cualquiera de ellos o la combinación de los mismos inicia el proceso.
El primer estímulo de senescencia conocido es el acortamiento de telómeros, debido a la ausencia de telomerasa en la mayor parte de las células somáticas. Durante la fase S de la replicación celular se pierde una sección del extremo de un cromosoma debido a la unidireccionalidad de las ADN polimerasas.[3] Por otra parte, estudios recientes han demostrado que la disfunción telomérica inicia la senescencia al activar una señal de daño genético persistente.[4]
Esta señal puede producirse en varios sitios no teloméricos del ADN de la célula, ya sea por el rompimiento de la doble hélice,[5] por la presencia de compuestos como los inhibidores de la histona deacetilasa, que relaja la cromatina sin inducir daño en el ADN, o bien por el supresor de tumores p53, que pueden activar la proteína ataxia telangiectasia mutada (ATM), que inicia la respuesta de daño genético.
Cuando se trabaja en condiciones in vitro, se observan fuentes diferentes de estrés que inducen senescencia, tales como los sustratos inadecuados, suero (la célula experimenta plasma in vivo, no suero), y estrés oxidativo (las condiciones atmosféricas son hiperfisiológicas).[6]La senescencia celular in vivo puede influir en las células de los tejidos circundantes y a nivel sistémico, al promover la inflamación.[7]
Fenotipo de la célula senescente[editar]
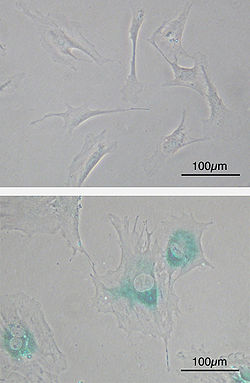
Aunque no todas las células senescentes presentan todas las características fenotípicas descritas a continuación, la presencia de cualquiera puede ser indicio suficiente para declarar el estado senescente.
- Aumento del tamaño de la célula, algunas veces hasta el doble del tamaño original. Esta primera característica de la senescencia celular fue descubierta por Hayflick.
- Aumento de la β-galactosidasa, producto de cambios en el compartimiento lisosomal, que aumenta su tamaño, lo que incrementa los niveles enzimáticos a un punto tal que la actividad de la β-galactosidasa puede ser detectada a pH 6.0 (subóptimo), y llega a niveles mucho más altos que los codificados regularmente por el gen GLB1,[8] la presencia de niveles aumentados de β-galactosidasa es detectada por tinción histoquímica en la mayor parte de las células.
- Expresión de inhibidores kinasas ciclin-dependientes (CDKi, en inglés) p16INK4a y p21WAF1: son capaces de activar procesos de senescencia sin activar la respuesta de daño genético común al proceso.[9] Estos inhibidores son parte de las rutas de supresión de tumores asociadas a las proteínas p53 y a la proteína de retinoblastoma (pRB), que crean focos de heterocromatina asociados a la senescencia (SAHF, en inglés), lo que silencia genes pro-proliferación.[10] Estos inhibidores aumentan en cantidad con el paso de los años y se ha visto que están vinculados con la disminución de células progenitoras en múltiples tejidos. Es importante hacer notar que la mayor parte de las células cancerígenas poseen defectos en las rutas mencionadas (pRB y p53), las células senescentes pueden identificarse fácilmente por medio de las proteínas HIRA y HP1β, que se observan exclusivamente en SAHF y que no están asociadas a ninguna otra patología.
- El ADN de células senescentes también presenta alteraciones en la cromatina que refuerzan la senescencia (SCARS). Estos focos tiene proteínas de daño persistente activadas como las fosfo-ATM y ataxia telangiectasia, y se pueden distinguir de los focos de daños transitorios y habitualmente incluyen telómeros disfuncionales.
Senescencia y envejecimiento[editar]
Existe una fuerte relación entre la senescencia celular y el envejecimiento. Varios estudios se han centrado especialmente en el papel de la proteína p53. En pruebas en ratones que fueron modificados para producir niveles altos de p53 se encontraron dos resultados muy disímiles: un grupo presentaba tasas de cáncer casi inexistentes, ya que p53 es un supresor de tumores, pero su vida media era significativamente más corta y presentaban síntomas de envejecimiento a edades tempranas. Esto llevó a determinar la correlación entre las altas tasas de senescencia celular y el envejecimiento,[11] pero, de manera paralela, algunos ratones que tenían una modificación que hacía que los niveles de p53 solo fueran más altos después de la activación natural de la respuesta de senescencia, tenían la misma tasa de resistencia al cáncer, pero una longitud de vida normal, las proteínas p53 reguladas naturalmente interactuaban con un efecto opuesto a las no reguladas,[12] lo que mostró la influencia de otros factores fisiológicos sobre los procesos de senescencia celular.
Restricción calórica y senescencia (envejecimiento)[editar]
Distintos factores fisiológicos pueden afectar las tasas de envejecimiento de un ser vivo. La restricción calórica es una de ellas, al limitar la cantidad de calorías disponibles sin caer en la desnutrición. Distintos modelos han aumentado su expectativa de vida promedio,[13] Este proceso parece estar asociado a la actividad de genes reguladores de información silenciosa, o sirtuinas, que codifican la producción de deacetilasas NAD dependientes en formas de vida menores como C. elegans y que pueden tener formas homólogas en mamíferos.
Se determinó que ortólogos de sirtuina 2 son necesarios para que la restricción calórica tenga un efecto de aumento de longevidad en C. elegans, y su ortólogo en mamíferos, la sirtuina T1, se encuentra en mayor cantidad en tejidos de mamíferos con restricción calórica. La relación aún no se ha confirmado en mamíferos.[14] La hipótesis principal detrás de esta correlación fue del laboratorio Guarente, donde se postula que en condiciones de restricción dietaria existe un cambio metabólico de fermentación a respiración,[15] lo que aumenta los niveles de NAD+ intracelular, lo cual aumentaría la actividad de la sirtuina 2. Sin embargo, esta hipótesis haría imposible el aumento de la expectativa de vida de organismos que sean incapaces de llevar a cabo ese cambio metabólico.
Referencias[editar]
- ↑ Hayflick, L. (Marzo de 1965). «The limited in vitro lifetime of human diploid cell strains». Experimental Cell Research 37: 614-636. ISSN 0014-4827. PMID 14315085. doi:10.1016/0014-4827(65)90211-9. Consultado el 21 de marzo de 2023.
- ↑ Shay, J. W.; Wright, W. E. (Octubre de 2000). «Hayflick, his limit, and cellular ageing». Nature Reviews. Molecular Cell Biology 1 (1): 72-76. ISSN 1471-0072. PMID 11413492. doi:10.1038/35036093. Consultado el 21 de marzo de 2023.
- ↑ Campisi, Judith (25 de febrero de 2005). «Senescent cells, tumor suppression, and organismal aging: good citizens, bad neighbors». Cell 120 (4): 513-522. ISSN 0092-8674. PMID 15734683. doi:10.1016/j.cell.2005.02.003. Consultado el 21 de marzo de 2023.
- ↑ Nakamura, Asako J.; Chiang, Y. Jeffrey; Hathcock, Karen S.; Horikawa, Izumi; Sedelnikova, Olga A.; Hodes, Richard J.; Bonner, William M. (3 de noviembre de 2008). «Both telomeric and non-telomeric DNA damage are determinants of mammalian cellular senescence». Epigenetics & Chromatin 1 (1): 6. ISSN 1756-8935. PMC 2584625. PMID 19014415. doi:10.1186/1756-8935-1-6. Consultado el 21 de marzo de 2023.
- ↑ Di Leonardo, A.; Linke, S. P.; Clarkin, K.; Wahl, G. M. (1 de noviembre de 1994). «DNA damage triggers a prolonged p53-dependent G1 arrest and long-term induction of Cip1 in normal human fibroblasts». Genes & Development 8 (21): 2540-2551. ISSN 0890-9369. PMID 7958916. doi:10.1101/gad.8.21.2540. Consultado el 21 de marzo de 2023.
- ↑ Parrinello, Simona; Samper, Enrique; Krtolica, Ana; Goldstein, Joshua; Melov, Simon; Campisi, Judith (Agosto de 2003). «Oxygen sensitivity severely limits the replicative lifespan of murine fibroblasts». Nature Cell Biology 5 (8): 741-747. ISSN 1465-7392. PMC 4940195. PMID 12855956. doi:10.1038/ncb1024. Consultado el 21 de marzo de 2023.
- ↑ Lyamina, S.; Baranovskii, D.; Kozhevnikova, E.; Ivanova, T.; Kalish, S.; Sadekov, T.; Klabukov, I.; Maev, I. et al. (2023). «Mesenchymal Stromal Cells as a Driver of Inflammaging». International Journal of Molecular Sciences 24 (7): 6372. ISSN 1422-0067. PMC 10094085. PMID 37047346. doi:10.3390/ijms24076372. Consultado el 28 de marzo de 2024.
- ↑ Lee, Bo Yun; Han, Jung A.; Im, Jun Sub; Morrone, Amelia; Johung, Kimberly; Goodwin, Edward C.; Kleijer, Wim J.; DiMaio, Daniel et al. (Abril de 2006). «Senescence-associated beta-galactosidase is lysosomal beta-galactosidase». Aging Cell 5 (2): 187-195. ISSN 1474-9718. PMID 16626397. doi:10.1111/j.1474-9726.2006.00199.x. Consultado el 21 de marzo de 2023.
- ↑ Rodier, Francis; Coppé, Jean-Philippe; Patil, Christopher K.; Hoeijmakers, Wieteke A. M.; Muñoz, Denise P.; Raza, Saba R.; Freund, Adam; Campeau, Eric et al. (Agosto de 2009). «Persistent DNA damage signalling triggers senescence-associated inflammatory cytokine secretion». Nature Cell Biology 11 (8): 973-979. ISSN 1476-4679. PMC 2743561. PMID 19597488. doi:10.1038/ncb1909. Consultado el 21 de marzo de 2023.
- ↑ Narita, Masashi; Nũnez, Sabrina; Heard, Edith; Narita, Masako; Lin, Athena W.; Hearn, Stephen A.; Spector, David L.; Hannon, Gregory J. et al. (13 de junio de 2003). «Rb-mediated heterochromatin formation and silencing of E2F target genes during cellular senescence». Cell 113 (6): 703-716. ISSN 0092-8674. PMID 12809602. doi:10.1016/s0092-8674(03)00401-x. Consultado el 21 de marzo de 2023.
- ↑ Hinkal, George W.; Gatza, Catherine E.; Parikh, Neha; Donehower, Lawrence A. (Abril de 2009). «Altered senescence, apoptosis, and DNA damage response in a mutant p53 model of accelerated aging». Mechanisms of Ageing and Development 130 (4): 262-271. ISSN 1872-6216. PMC 2722837. PMID 19396980. doi:10.1016/j.mad.2009.01.001. Consultado el 21 de marzo de 2023.
- ↑ de Keizer, Peter L. J.; Laberge, Rémi-Martin; Campisi, Judith (Julio de 2010). «p53: Pro-aging or pro-longevity?». Aging 2 (7): 377-379. ISSN 1945-4589. PMC 2933881. PMID 20657035. doi:10.18632/aging.100178. Consultado el 21 de marzo de 2023.
- ↑ Colman, Ricki J.; Anderson, Rozalyn M.; Johnson, Sterling C.; Kastman, Erik K.; Kosmatka, Kristopher J.; Beasley, T. Mark; Allison, David B.; Cruzen, Christina et al. (10 de julio de 2009). «Caloric restriction delays disease onset and mortality in rhesus monkeys». Science (New York, N.Y.) 325 (5937): 201-204. ISSN 1095-9203. PMC 2812811. PMID 19590001. doi:10.1126/science.1173635. Consultado el 21 de marzo de 2023.
- ↑ Cohen, Haim Y.; Miller, Christine; Bitterman, Kevin J.; Wall, Nathan R.; Hekking, Brian; Kessler, Benedikt; Howitz, Konrad T.; Gorospe, Myriam et al. (16 de julio de 2004). «Calorie restriction promotes mammalian cell survival by inducing the SIRT1 deacetylase». Science (New York, N.Y.) 305 (5682): 390-392. ISSN 1095-9203. PMID 15205477. doi:10.1126/science.1099196. Consultado el 21 de marzo de 2023.
- ↑ Lin, Su-Ju; Kaeberlein, Matt; Andalis, Alex A.; Sturtz, Lori A.; Defossez, Pierre-Antoine; Culotta, Valeria C.; Fink, Gerald R.; Guarente, Leonard (18 de julio de 2002). «Calorie restriction extends Saccharomyces cerevisiae lifespan by increasing respiration». Nature 418 (6895): 344-348. ISSN 0028-0836. PMID 12124627. doi:10.1038/nature00829. Consultado el 21 de marzo de 2023.
